Protein-Protein
Protein-Ligand
構造アライメント その他
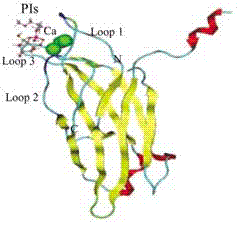
カルシウムイオン存在・非存在化でのフォスファチジルイノシトール4,5-ビスホスフェイトの結合性を検討
結合部位を検索し、restraintをかけてエネルギーの極小化(カルシウムイオンの結合構造を予測)
→リガンドドッキングシミュレーション
ドッキングシミュレーションの練習
DNAポリメラーゼIII(1JQL.pdb)は既にアクセサリー蛋白と結合した構造が解析されている。シミュレーションにより再現できるか。
結果:
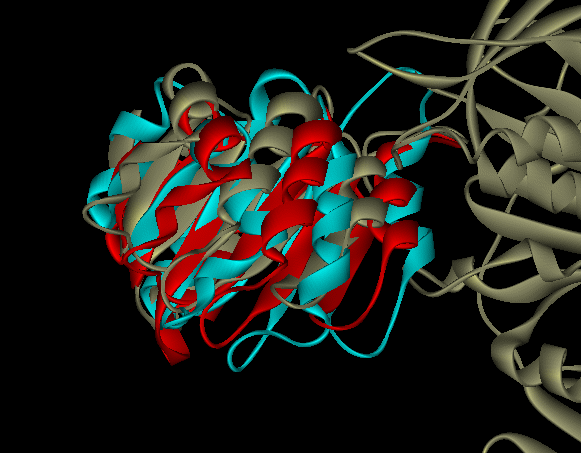
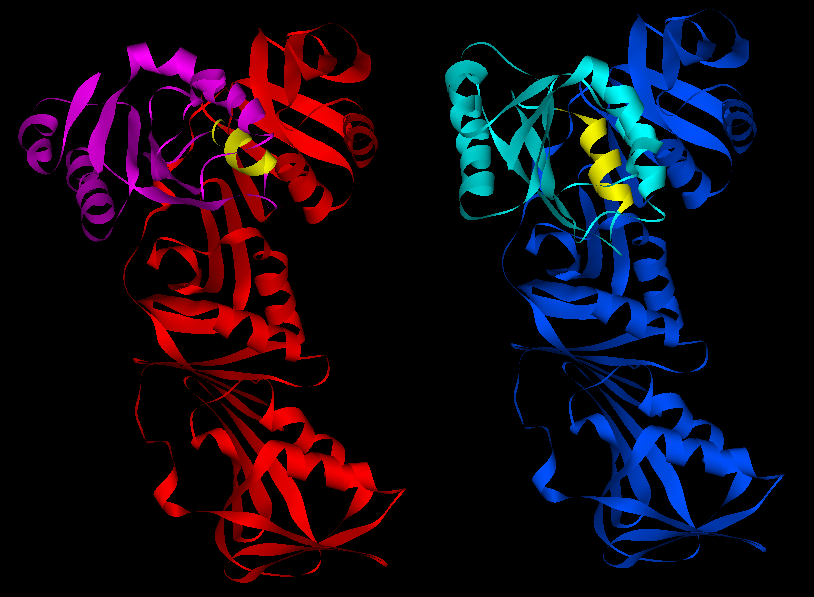
赤:DOT 水色:ZDOCK Original Ptrediction
MOEを使ったドッキングシミュレーションの基礎
PDBファイルの取得
PDBファイルの最適化
1. MOEを起動する。
2. File/Openで開く。
PDB fileの場合、ファイルを選択後、右のForce TypeをPDBにしてOKを押す。その後は2度OKを押す。
3. ウィンドウ右上ボタンの2つめ、Sequence Editorを開く。
4. Sequence EditorウィンドウのChainの項目が1つでない場合、H2O、リガンド等のchainを押し、Edit/Delete Selected Chainで削除する。
5. Select/Element/Heavy Atomを選ぶ。
6. Edit/Fixを選ぶ。
7. Compute/Energy Minimizeを選ぶ。左上に数字が現れ、しばらくすると終了する。
8.File/Saveでファイル名を指定して保存する。ファイル属性を.moeとしておくとわかりやすい。
(後で使うのでポテンシャルエネルギーを記録する。)
リガンドの取得
a.ブルーバックスの「パソコンで見る動く分子辞典」本間善夫・川端潤の附属CDを参照する。
b.インターネットで得る。
c.SciFinder Scholarを利用する。
d.化合物データベースから得る。
e.自分でbuildして最適化する。
ドッキングシミュレーション
1. MOEを起動する。
2. File/Openで開く。
3. ウィンドウ右上ボタンの2つめ、Sequence Editorを開く。
4.リガンドのchainを選択肢マウス右クリックでAtoms/Select
5.MOEメニューの下の入力欄にASEDock[]と入力してリターン。
計算終了まで待つ。
検討の仕方
mdbというファイルが作成され、いくつかの候補とその状態が表示されるのでそれぞれの項目について検討する。相互作用エネルギーを知りたいときはリガンド分子を選択し、MOE/Compute/Ptential energyとすると画面上部に表示される。
Last update 2018/09/04